数据驱动的细胞培养基优化:机器学习助力生物医学进步
发布时间:2025-06-09 浏览次数:320 分享:
1. 研究背景与挑战
核心问题:哺乳动物细胞培养中,培养基成分的复杂互作导致传统优化方法(如实验设计DoE)效率低下。
现有局限:经验依赖性强,易受人为偏差影响;难以平衡细胞生长速率(96小时)与最终产量(168小时)。
技术机遇:主动机器学习(Active ML)可通过“预测-验证”闭环减少实验量,实现多参数同步优化。
2. 创新方法:从传统DoE到智能ML

本研究技术路线如图1)如所示:
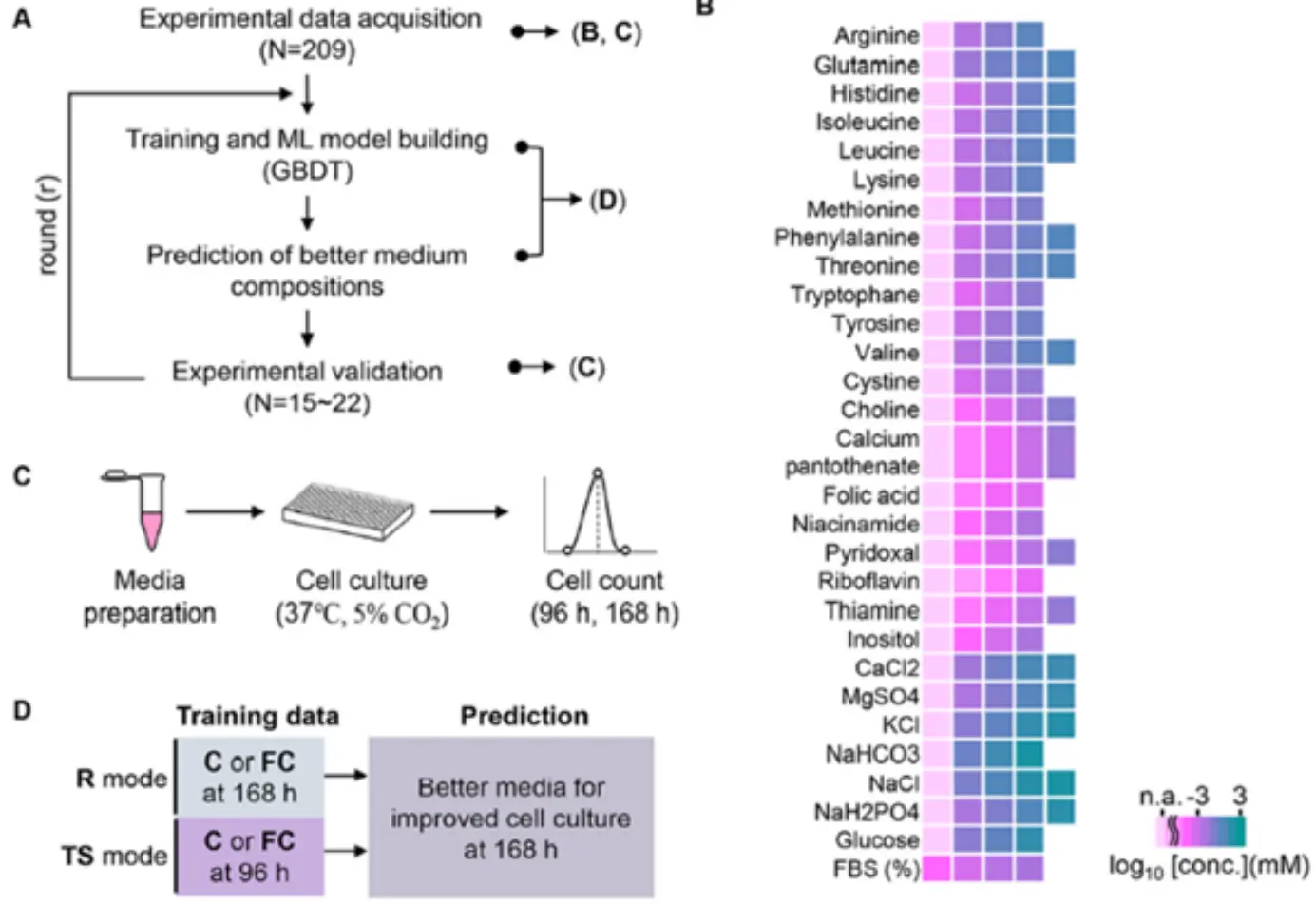
图1 技术路线图
1)模型构建:
细胞模型:HeLa-S3细胞(人宫颈癌细胞系)
基础培养基:EMEM(含31种成分)
算法:梯度提升决策树(GBDT)
2)双模式优化:
常规模式(R):基于168小时细胞浓度数据优化最终产量
节时模式(TS):基于96小时数据预测168小时产量(利用早期-晚期浓度相关性)
3)四轮主动学习:
初始生成209种梯度培养基
每轮筛选15-22种高潜力培养基实验验证
迭代更新训练集(共503组数据)
3. 关键结果
1)培养基优化效能
细胞浓度提升:
R模式:11种培养基168小时浓度显著高于EMEM(最高提高50%)
TS模式:10种培养基96小时浓度提升,但仅30%维持至168小时
模型精度:
四轮迭代后预测误差(RMSE)降低40%( p<0.05)
数据标准化(Fold Change, FC)较原始数据(C)提升模型鲁棒性
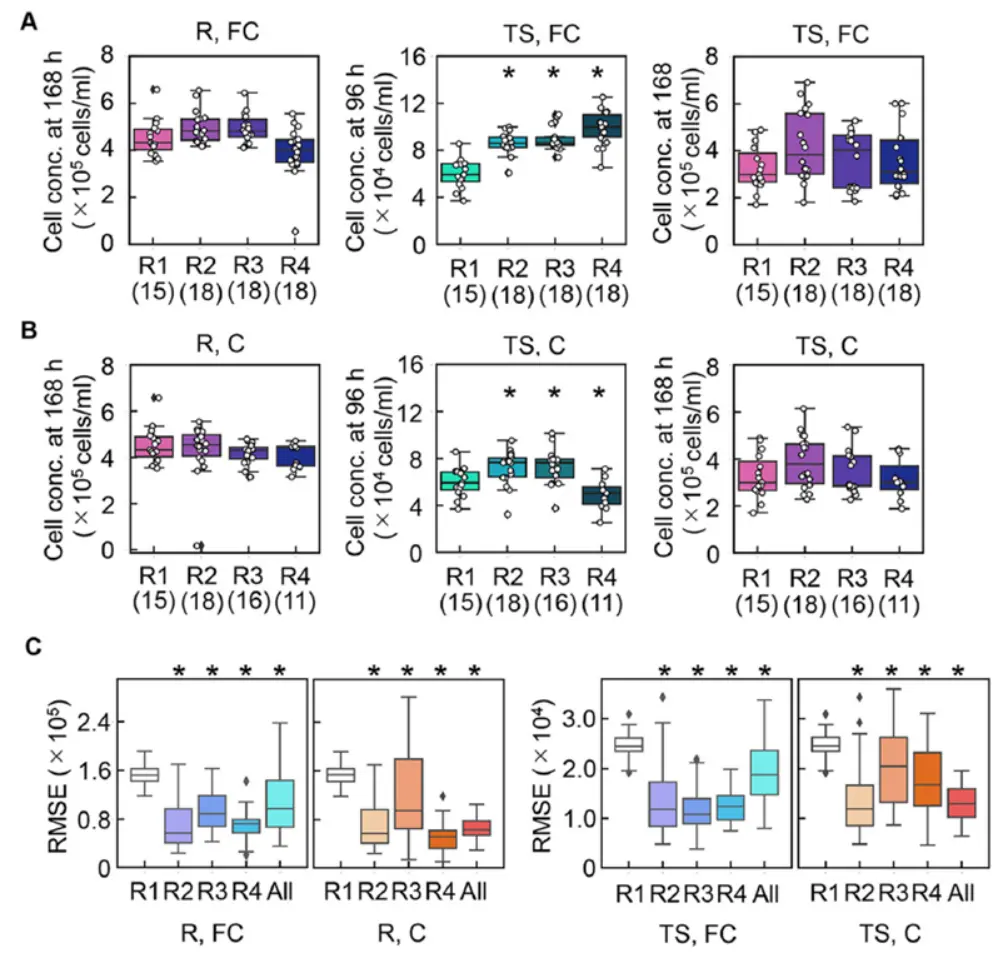
图2 R/TS模式效能对比
2)关键成分鉴定
特征重要性分析:9种成分贡献80%生长影响(如谷氨酰胺升高、半胱氨酸降低)
时序差异:甲硫氨酸仅晚期(168小时)关键
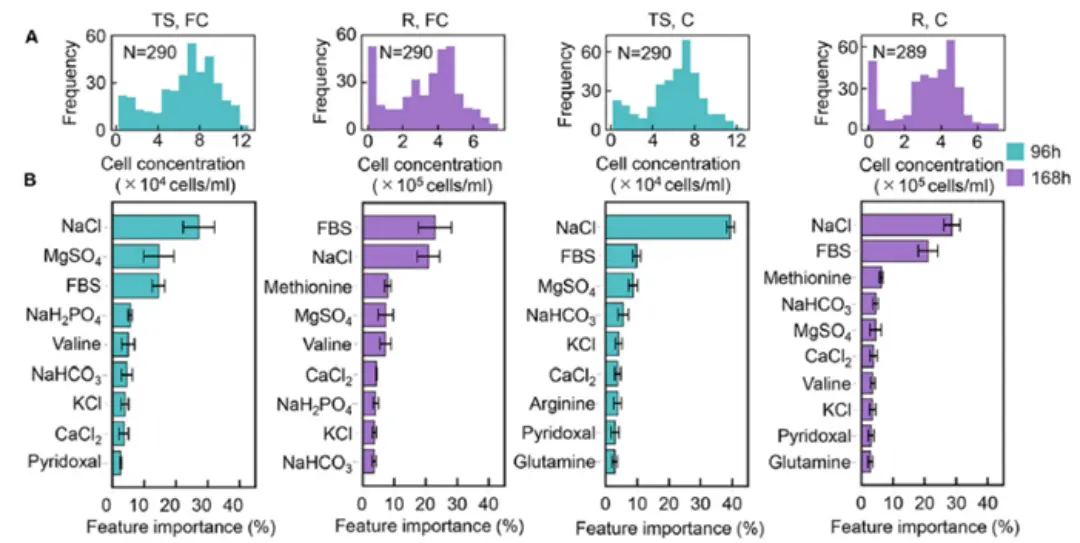
图3 培养基成分对细胞培养的关键影响
A.不同模型下细胞培养产量的分布;B.重要性排名前9的成分(累积贡献80%)
规模放大验证:
最优培养基R445:168小时浓度提高50%(p <0.01)
成分共性:R445与失效培养基TS471均缺失磷酸氢钠和硫胺素
3) 转录组机制解析
基因表达重编程:
R445激活增殖通路(如细胞发育相关GO term)
TS471引发表达紊乱(差异基因数↑100倍)
功能富集:
R445特异性富集:细胞周期调控基因(如mTORC1通路)
EMEM富集:代谢调控基因
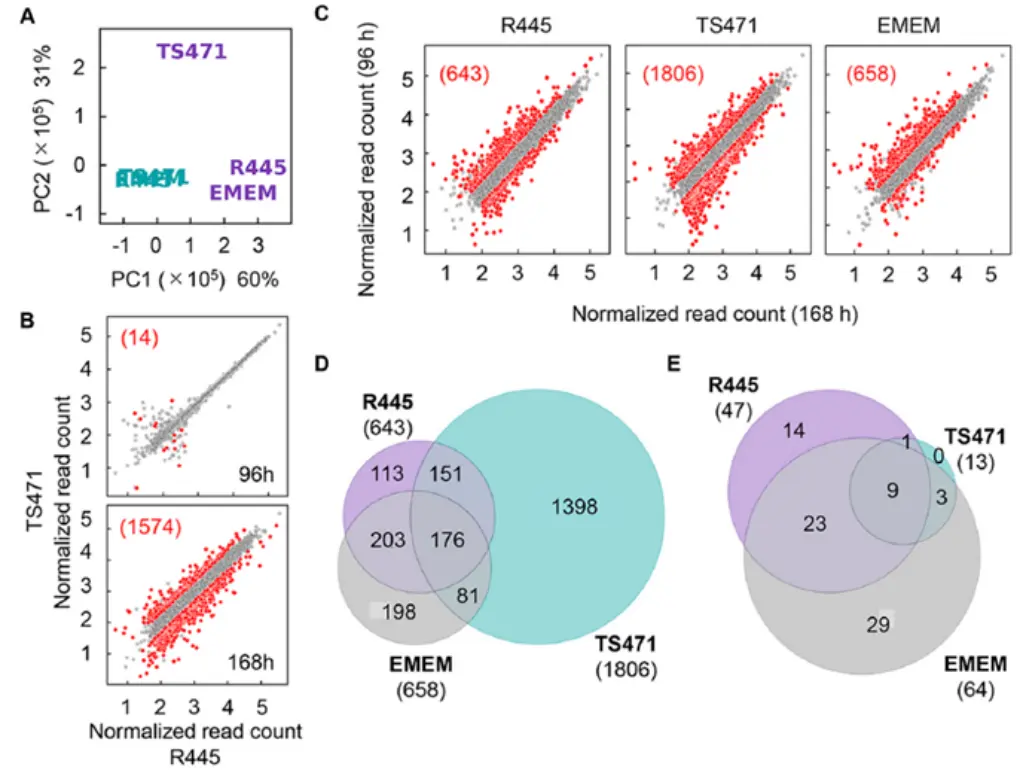
图4 基因表达变化与培养基优化的关联性研究
4. 讨论与局限
1)模式对比
R模式:稳定提升最终产量(C1簇占比提高)
TS模式:节省50%实验时间,但33%培养基因“生长-产量权衡”失效(C2簇)
2)技术突破
数据驱动闭环:首次整合ML预测→实验验证→转录组反馈
节时策略:利用96/168小时浓度相关性(r=0.82)加速优化
3)局限与展望
细胞普适性:当前仅验证HeLa细胞,需拓展至原代/干细胞
血清依赖:含FBS培养基成分不透明,下一步开发无血清方案
算法升级:引入时间序列数据提升TS模式稳定性
5. 应用价值
生物制药:快速定制单抗生产培养基(成本降低30%)
再生医学:优化干细胞扩增体系
开源资源:
代码库:https://github.com/yuki020527/medium_optimization
RNA-seq数据:DRA017793 (DDBJ)
总之,本研究建立了主动机器学习驱动的培养基优化新范式,通过双模式迭代和转录组验证,突破传统方法瓶颈。其中四轮迭代减少60%实验量,并发现9种核心成分及时序调控规律,为生物制造与精准医疗提供工具包。
参考文献:Y Ozawa, T Hashizume, BW Ying. (2025). A data-driven approach for cell culture medium optimization. Biochemical Engineering Journal, 241, 109591. https://doi.org/10.1016/j.bej.2024.109591
来源:微生物安全与健康网,作者~陈诺。






